- Title: Clinical Knowledge Graph Construction and Evaluation with Multi-LLMs via Retrieval-Augmented Generation
- 저자: Udiptaman Das, Krishnasai B. Atmakuri, Duy Ho, Chi Lee, Yugyung Lee
의학적 서술문에서 정확하고 임상적으로 관련성이 있는 지식 그래프(KGs)를 구축하는 것은 생물의학 정보학에서 근본적인 도전 과제입니다. 임상 KG는 설명 가능한 AI, 의사결정 지원, 그리고 환자의 장기적 모델링을 가능하게 하지만, 전통적인 접근법은 여전히 제약되어 있습니다. 이 논문에서는 free-text에서 직접 KG를 구축하고 평가하는 최초의 end-to-end 프레임워크를 소개합니다. 우리의 파이프라인은 지속적인 정교화와 자기 감독 평가를 지원하여, 높은 정확도의 구성과 시간에 따른 동적 그래프 개선을 가능하게 합니다.
1. **첫 번째 기여**는 multi-agent 프롬팅, LLM 기반 정교화, 그리고 golden-standard 라벨 없이 지속적인 평가를 통합한 KG-RAG(지식 그래프-추출증강생성) 프레임워크입니다. 이는 마치 여러 전문가들이 함께 작업하여 복잡한 건물을 설계하고 검토하는 것과 같습니다.
2. **두 번째 기여**는 schema에 제약을 받고 FHIR와 일치하는 EAV 추출 모듈, 그리고 ontology 매핑 및 OWL 기반 인코딩입니다. 이를 통해 우리는 마치 다양한 도구를 사용해 정확한 건축도면을 만드는 것처럼, 복잡한 의학적 정보를 구조화할 수 있습니다.
3. **세 번째 기여**는 암 서술문에서 사실성, 의미 체계의 뿌리 깊은 연결, 그리고 관계의 완전성을 평가하는 방법을 경험적으로 검증합니다. 이는 마치 의사가 복잡한 환자 데이터를 분석하고 정확한 진단을 내리는 것과 같습니다.
# 소개
비구조화된 의학적 서술문에서 정확하고 임상적으로 관련성이 있는 지식 그래프(KGs)를 구축하는 것은 생물의학 정보학에서 근본적인 도전 과제입니다. 임상 KG는 설명 가능한 AI, 의사결정 지원 및 환자 장기 모델링을 가능하게 하지만, 전통적인 접근법은 여전히 제약되어 있습니다. FHIR와 같은 경직된 스키마는 의미적 유연성을 갖추지 못하고, SNOMED CT, LOINC, RxNorm과 같은 온톨로지는 표준 용어를 제공하지만 암 등 복잡한 영역에서 필요한 시간적, 맥락적, 추론적 미묘함을 포착하는 데 어려움이 있습니다.
통상적인 규칙 기반 또는 수작업 KG 구축 파이프라인은 단단하고 확장하기 어렵으며 임상 서술문의 진화하는 언어와 구조에 적합하지 않습니다. 이에 반해, 대형 언어 모델(LLMs)은 의미 해석, 관계 탐색 및 맥락 인식 생성을 위한 강력한 도구로 부상했습니다. Gemini 2.0 Flash, GPT-4o, 그리고 Grok 3와 같은 모델들은 텍스트로부터 KG를 자동화된 방식으로 구축하는 데 잠재력을 보여주고 있지만, 그들의 출력은 환영, 의미 유변, 사실적 일관성 부족 등과 같은 문제에 취약합니다. 이러한 이슈는 특히 암과 같이 고스테이크 영역에서 중요합니다.
최근의 연구들인 CancerKG.ORG, EMERGE, 그리고 CLR2G는 LLM을 구조화되거나 다중 모달 소스와 통합하는 것을 탐색하고 있지만, 임상 텍스트로부터 직접 KG를 구성하고 검증하기 위한 일반화 가능한 파이프라인은 부족합니다.
본 논문에서는 multi-agent 프롬팅과 그래프 기반 추출 증강 생성(KG-RAG) 접근법을 사용하여 free-text에서 임상 지식 그래프를 구축하고 평가하는 최초의 end-to-end 프레임워크를 소개합니다. 우리의 파이프라인은 지속적인 정교화와 자기 감독 평가를 지원하여 높은 정확도의 구성과 시간에 따른 동적 그래프 개선을 가능하게 합니다.
우리는 다음의 장점을 갖춘 multi-agent LLM 파이프라인을 활용합니다:
-
Gemini 2.0 Flash는 스키마 지향 Entity–Attribute–Value (EAV) 추출을 위해 사용됩니다;
-
GPT-4o는 맥락적 풍부화, 온톨로지 정렬 및 반영 기반 정교화를 위해 사용됩니다;
-
Grok 3은 모순 시험과 보수적인 필터링을 통해 검증에 사용됩니다.
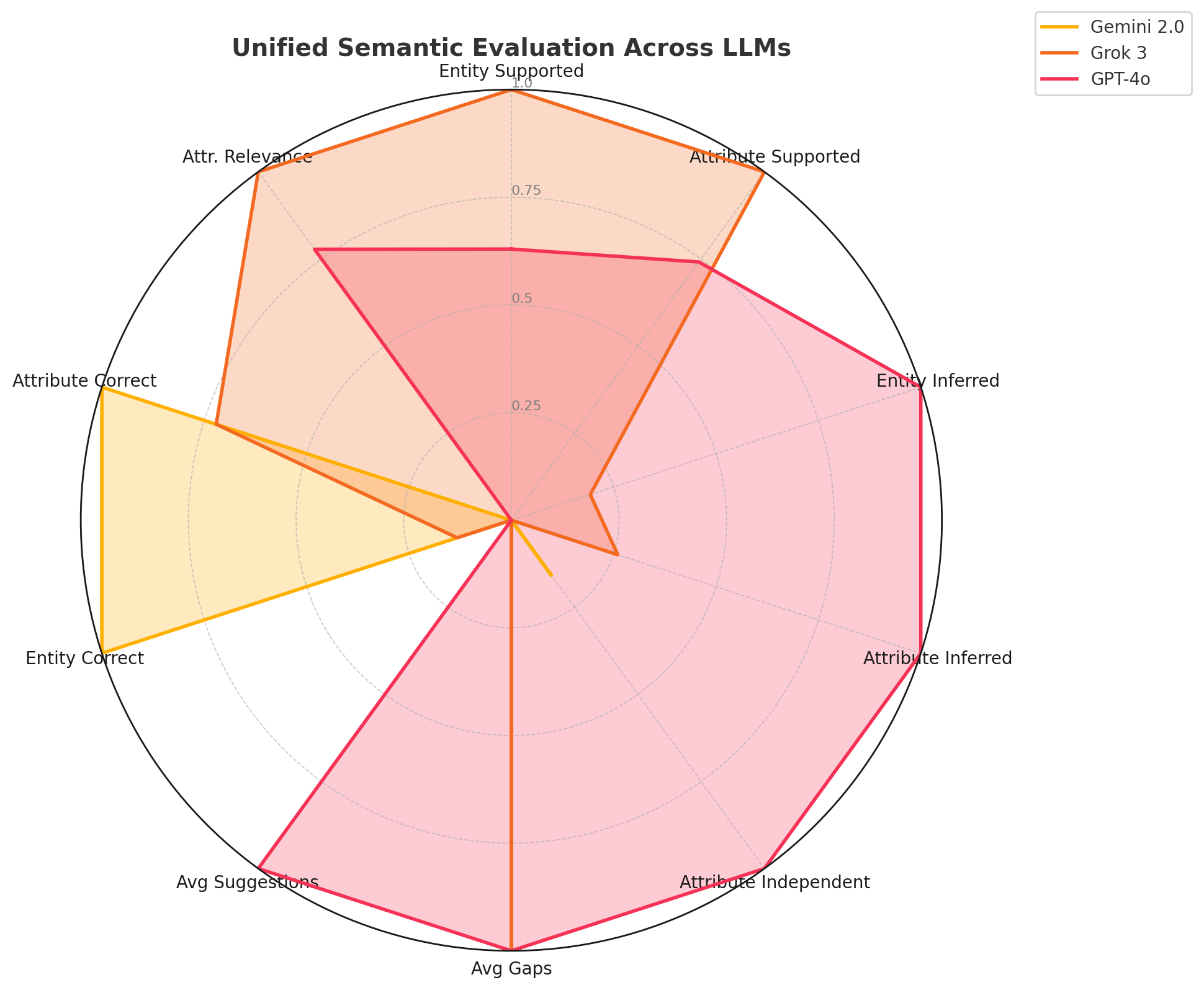
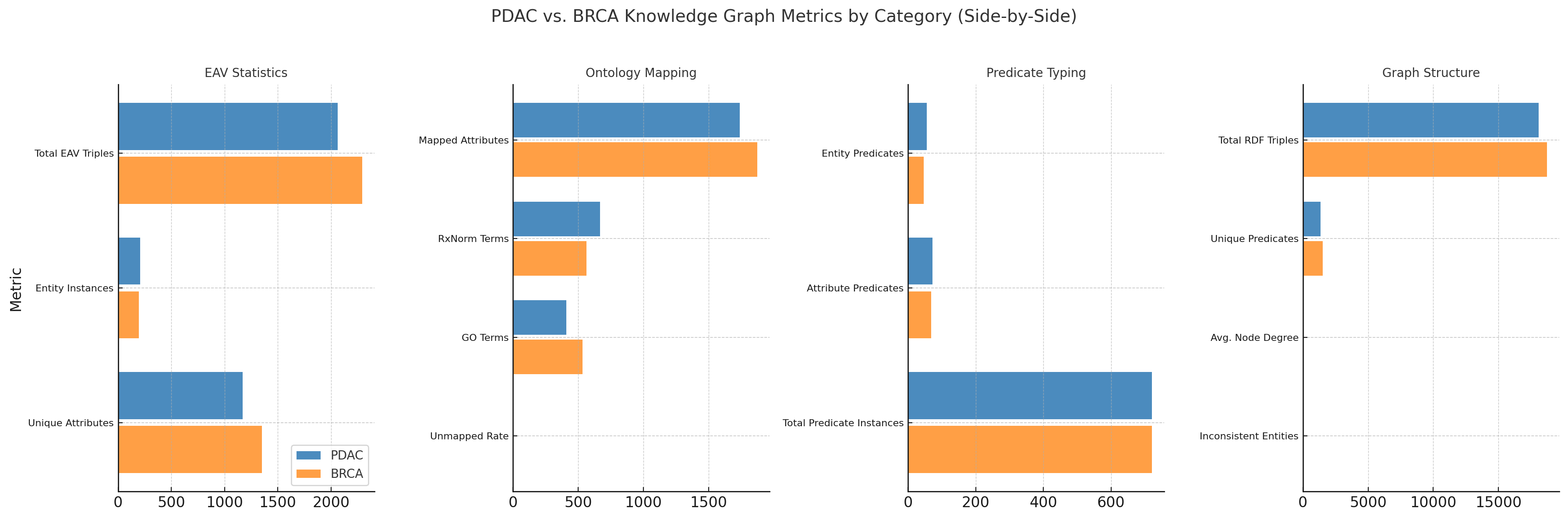
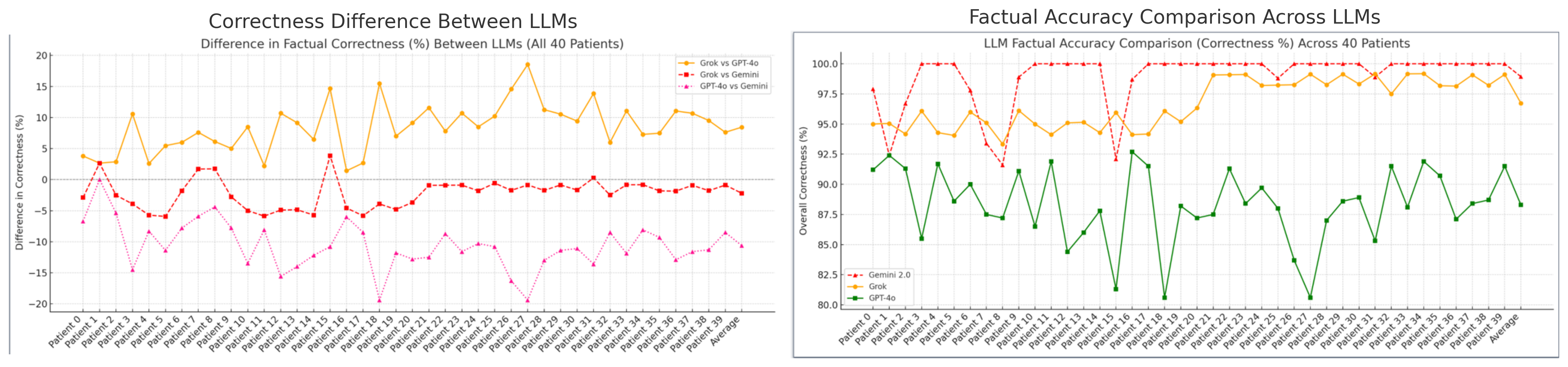
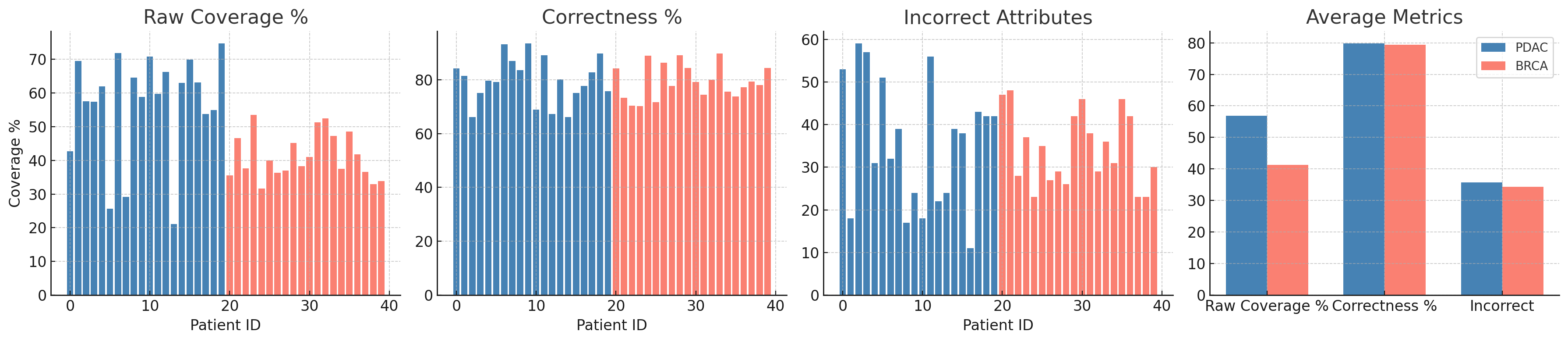
모든 트리플은 SNOMED CT, LOINC, RxNorm, GO, ICD로 매핑되어 RDF/RDFS/OWL에서 의미적 추론을 위해 인코딩됩니다. 신뢰도 지표는 모델의 동일성과 생물 의학 온톨로지와의 일치로부터 도출됩니다. 우리는 CORAL 데이터셋에서 40개의 임상 암 보고서를 대상으로 우리의 방법을 시연하고, 트리플 정확도, 온톨로지 커버리지, 관계 다양성 및 그래프 연결성을 평가합니다.
우리의 주요 기여는 다음과 같습니다.
- 우리는 golden-standard 라벨 없이 multi-agent 프롬팅, LLM 기반 정교화 및 지속적인 평가를 통합한 KG-RAG 프레임워크를 제안합니다.
- 우리는 스키마에 제약을 받고 FHIR와 일치하는 EAV 추출 모듈을 소개하며, 이는 온톨로지 매핑과 OWL 기반 인코딩을 포함합니다.
- 우리는 암 서술문에서 사실성, 의미 체계의 뿌리 깊은 연결, 그리고 관계의 완전성을 평가하는 방법을 경험적으로 검증합니다.
다양한 LLM 공감대, 온톨로지 지향 및 반복적 정교화를 통합함으로써 우리의 시스템은 임상 KG 구축에 대해 확장 가능하고 설명 가능하며 검증 가능한 접근 방식을 제공하여 실시간 의사결정 지원과 차세대 임상 AI의 기초를 마련합니다.
관련 연구
하이브리드 지식 그래프-언어 모델(KG–LLM) 시스템, 추출 증강 생성(RAG), 그리고 대조 학습의 최근 진전은 임상 정보학에 큰 영향을 미쳤습니다. 그러나 많은 기존 접근법은 사전 정의된 스키마, 큐레이션된 코퍼스 또는 다중 모달 데이터를 의존하고 있습니다. 우리의 연구는 중요한 공백을 해결합니다: 구조화되지 않은 임상 서술문으로부터 직접 추론 가능한 지식 그래프를 구성하는 방법입니다.
스키마 제약 KG–LLM 하이브리드
CancerKG.ORG 은 GPT-4, LLaMA-2, FLAN-T5와 같은 대형 언어 모델(LLMs)을 큐레이션된 대장암 지식과 통합합니다. 또한 구조화된 메타 프로필 및 가드레일 RAG를 사용합니다. KnowGL, KnowGPT, 그리고 KALA 와 같은 다른 프레임워크는 지식 그래프 정렬의 효능을 입증하지만, 정적 온톨로지를 의존하고 있습니다. 이에 반해 우리의 시스템은 자동적인 엔티티-특성-값(EAV) 추출, 확률적 트리플 스코어링 및 반복적 온톨로지 정렬을 통해 스키마의 동적 진화를 가능하게 하여 수작업 큐레이션 또는 고정된 스키마가 필요하지 않습니다.
다중 모달 RAG와 예측 시스템
EMERGE, MedRAG, 그리고 GatorTron-RAG 와 같은 시스템은 임상 노트, 구조화된 전자 건강 기록(EHR) 데이터 및 생물의학 그래프를 진단 또는 위험 분류를 위한 예측 작업에 결합합니다. 이러한 모델들은 분류에 효과적이지만 심볼적 지식 표현에는 집중하지 않습니다. 우리의 접근법은 완전히 텍스트 기반 파이프라인을 사용하여 설명 가능한, 심볼적 트리플 구성을 강조하며, 외부 모달성 또는 고정된 작업 목표에 대한 의존성을 제거합니다.
대조 및 신뢰 인식 LLM 생성
Reflexion, TruthfulQA와 같은 방법을 통해 신뢰 인식 LLM 생성이 탐색되었습니다. 이러한 방법은 자반성과 환영 감소 기술을 사용합니다. 마찬가지로 CLR2G는 방사선 보고서 생성을 위한 다중 모달 대조 학습을 적용합니다. 이러한 접근법들은 의미적 제어를 강화하지만, 우리의 프레임워크는 Gemini, GPT-4o 및 Grok과 같은 여러 LLM에 대한 트리플 스코어링, 엔트로피 인식 신뢰 필터링 및 반영 기반 검증을 도입하여 특히 임상 텍스트에서 심볼적 추론에 집중합니다.
구조화된 추출과 표 중심 지식 그래프
WebLens, Hybrid.JSON, 그리고 COVIDKG와 같은 이전 플랫폼은 다양한 임상 데이터 표의 확장 가능한 구조적 추출 및 메타데이터 모델링을 소개했습니다. CancerKG는 구조화된 암 데이터베이스에 대한 수직 및 수평 메타데이터 모델링을 통해 이 방향을 확장합니다. 우리의 작업은 모달성과 범위에서 근본적으로 다릅니다: 표 정렬 또는 속성 정규화를 의존하지 않고, 프롬프트 기반 분해, 확률적 스코어링 및 온톨로지 지원 추론을 사용하여 free-text 보고서로부터 진화하는 임상 지식 그래프를 직접 구축합니다.
요약하면, 기존 시스템은 구조화된 지식 추출과 LLM-KG 통합에 중요한 기초를 마련했습니다. 그러나 우리의 기여는 구조체계의 제약을 없애고 multi-LLM 합의를 활용하여 사실적 신뢰성을 제공하는 자동 평가, 스키마 유연성 파이프라인입니다. 이를 통해 확장 가능하고 설명 가능하며 임상적으로 관련성이 있는 지식 그래프 구축이 가능하게 되며 새로운 도메인과 문서 유형에 동적으로 적응할 수 있습니다.
방법론
다중 에이전트 KG 구성 및 평가 파이프라인 개요
우리는 암 서술문에서 직접 임상적으로 검증 가능한, 의미적 상호 운용성이 있는 지식 그래프(KGs)를 구축하기 위한 multi-agent 프레임워크를 제안합니다. 이 시스템은 세 가지 최첨단 대형 언어 모델—Gemini 2.0 Flash, GPT-4.o, 그리고 Grok 3—을 조화롭게 관리하여 다섯 개의 모듈러 단계에서 각각 전문적인 역할을 부여합니다. 이 아키텍처는 구조화된, 설명 가능한, 온톨로지 정렬 지식 그래프를 비구조화된 임상 텍스트로부터 생성하도록 설계되었습니다.
-
EAV 추출 (Gemini 2.0 Flash): 특별히 제작된 프롬트와 FHIR 인식 템플릿을 사용하여 Gemini 2.0 Flash는 free-text 서술문에서 Entity–Attribute–Value (EAV) 트리플을 추출합니다. 추출된 엔티티들은 캐논적 임상 리소스 유형에 링크됩니다.
-
온톨로지 매핑 (Gemini 2.0 Flash): 속성과 값은 SNOMED CT, LOINC, RxNorm와 같은 표준 생물의학 사전어휘로 매핑됩니다. 이 단계는 용어를 정규화하고 임상 개념 간 의미적 일관성을 유지합니다.
-
관계 탐색 (Gemini 2.0 Flash + GPT-4.o): 엔티티와 속성 사이의 의미적 관계는 프롬프트 기반 추출 및 정교화를 통해 식별됩니다. Gemini 2.0 Flash는 후보 링크를 생성하고, GPT-4.o는 맥락 이해 및 온톨로지 정렬을 사용하여 관계 유형을 정제하고 검증합니다.
-
세미안틱 웹 인코딩 (RDF/RDFS/OWL): 모든 트리플은 RDF, RDFS, OWL과 같은 세미안틱 웹 표준을 사용하여 인코딩됩니다. 이를 통해 기호적 추론, SPARQL을 통한 지식 그래프 쿼리 및 외부 온톨로지 기반 시스템과의 통합이 가능합니다.
-
KG 검증 (Gemini 2.0 Flash + GPT-4.o + Grok 3): 복합 신뢰 함수는 각 트리플의 신뢰성을 평가하기 위해 적용됩니다. 신뢰 점수는 다음과 같이 구성됩니다:
- Gemini 2.0 Flash에서의 자기 일관성 (재프롬핑을 통해),
- GPT-4.o에서의 의미적 뿌리 깊은 연결 (증거 검색 및 일관성 확인),
- Grok 3에서의 견고성 검증 (반사상 및 적대적 평가).
이 파이프라인은 raw 임상 서술문으로부터 스키마 유연성, 온톨로지 정보 제공 및 모델 검증을 통한 지식 그래프 구축을 지원합니다. 모듈형 아키텍처는 하류 추론, 쿼리 및 임상 의사결정 지원 시스템에 통합되며 추출된 정보의 추적 가능성과 설명 가능성을 유지합니다.
단계 1: FHIR-지향 EAV 추출
우리 파이프라인의 첫 번째 단계는 Entity–Attribute–Value (EAV) 트리플 형태로 구조화된 임상 지식을 추출하는 것입니다. 이는 Gemini 2.0 Flash를 사용한 스키마 지향 프롬핑을 통해 이루어지며, 서술문에서 문법적 단서와 FHIR (Fast Healthcare Interoperability Resources) 사양의 의미적 제약을 통합합니다. 엔티티들은 동시에 FHIR로 타입화되어 의미적 일관성 및 상호 운용성을 보장합니다.
트리플 추출.
임상 서술문 코퍼스를 $`\mathcal{D} = \{x_1, x_2, \ldots, x_N\}`$로 정의하고 각 $`x_i \in \mathcal{D}`$에 대해 Gemini 2.0 Flash에 맞춘 구조화된 프롬프트 $`\pi(x_i)`$를 생성합니다.
\mathcal{T}_i = f_{\theta}(\pi(x_i)) = \left\{(e_j, a_j, v_j)\right\}_{j=1}^{k}, \quad \forall i \in [1, N]
각 트리플 $`(e_j, a_j, v_j)`$는 다음과 같이 구성됩니다:
- $`e_j \in \mathcal{E}_{\text{FHIR}}`$: FHIR 타입화된 엔티티 (예: Procedure, Observation),
- $`a_j`$: 임상 속성,
- $`v_j`$: 서술문 기반 값.
FHIR 타이핑 함수.
엔티티들은 표준 리소스 유형으로의 하류 매핑을 보장하기 위해 $`\phi_{\text{FHIR}} : \mathcal{E} \rightarrow \mathcal{E}_{\text{FHIR}}`$라는 타입화 함수를 통해 정규화됩니다.
엔트로피 기반 값 신뢰도.
값 예측의 확신을 평가하기 위해 $`v_j`$의 서브토큰 분포 $`P(v_j) = \{p_1, \ldots, p_m\}`$에 대한 토큰 수준 엔트로피를 계산합니다:
H(v_j) = -\sum_{t=1}^{m} p_t \log p_t
값 $`H(v_j) > \delta`$ (임계값 $`\delta`$)인 경우 추가 검증 또는 다중 모델 필터링이 필요합니다.
예시 EAV 트리플.
예시는 다음과 같습니다:
(Procedure, performed_by, SurgicalOncologist)
(Observation, hasLabResult, CA 19-9)
(HER2 Status, determines, Trastuzumab Eligibility)
이 EAVs는 후속 단계에서 온톨로지 매핑, 관계 탐색 및 세미안틱 웹 인코딩의 기반이 됩니다.
단계 2: 온톨로지 매핑 및 스키마 구축
세mantic 추론과 상호 운용성을 가능하게 하기 위해 추출된 EAV 개념들은 LLM 지도 추출 및 유사성 정렬을 통해 표준화된 생물 의학 온톨로지에 매핑됩니다. Gemini 2.0 Flash는 이 단계를 조정하고 OWL/RDFS 호환 스키마를 생성합니다.
온톨로지 어휘.
우리는 다음과 같은 온톨로지 집합을 정의합니다:
\mathcal{O} = \{\text{SNOMED CT}, \text{LOINC}, \text{RxNorm}, \text{ICD}, \text{GO}\}
각 $`\mathcal{O}_i`$는 도메인 특화된 개념을 제공합니다, 예를 들어 SNOMED: Weight Loss, LOINC: CA 19-9, RxNorm: FOLFIRINOX.
개념 매핑.
원시 용어 $`\mathcal{C}_{\text{raw}} = \{c_1, \ldots, c_M\}`$에 대해 다음과 같은 매핑을 정의합니다:
\mu: \mathcal{C}_{\text{raw}} \rightarrow \mathcal{C}_{\text{mapped}} \subseteq \bigcup_i \mathcal{O}_i
스코어링은 어휘적 및 의미적 유사성을 사용하여 수행됩니다:
\text{Score}(c_i, o_j) = \alpha \cdot \text{sim}_{\text{lex}} + \beta \cdot \text{sim}_{\text{sem}}, \quad \alpha + \beta = 1
스키마 구축.
매핑된 개념들은 OWL/RDFS로 인코딩됩니다:
- 클래스 타이핑: $`o_j \in \mathcal{O}_{\text{SNOMED}}`$인 경우, $`o_j`$를 OWL
Class로 선언합니다.
- 속성 의미론:
\begin{align*}
\texttt{hasLabResult} &\sqsubseteq \texttt{ObjectProperty}, \\
\texttt{domain(hasLabResult)} &= \texttt{Observation}, \\
\texttt{range(hasLabResult)} &= \texttt{LabTest}
\end{align*}
- TBox 포함:
ElevatedCA19_9 $`\sqsubseteq`$ AbnormalTumorMarker.
영구 URI.
각 개념은 해결 가능한 URI를 받으며, 예를 들어 “Weight Loss”에 대해 http://snomed.info/id/267036007이 할당됩니다.
결과.
이 단계는 OWL 추론, SPARQL 쿼리 및 Linked Data 통합을 지원하며 형식적 일관성과 의미적 추적 가능성을 보장하는 온톨로지 정렬 스키마를 생성합니다.
단계 3: 관계 탐색
이 단계는 고립된 EAV 트리플을 넘어 진단 추론, 시간 의존성 및 치료 논리를 포착하는 유형화된 관계로 지식 그래프를 풍부하게 합니다. 구조화된 프롬핑과 다중 에이전트 검증 (Gemini 2.0 Flash, GPT-4.o, Grok 3)을 사용하여 의미적 관계를 탐색하고 필터링합니다.
관계 타이핑: 각 관계 $`r_i \in \mathcal{R}`$는 다음과 같이 분류됩니다:
r_i \in
\begin{cases}
\mathcal{R}_{EE} & \text{(Entity–Entity)} \\
\mathcal{R}_{EA} & \text{(Entity–Attribute)} \\
\mathcal{R}_{AA} & \text{(Attribute–Attribute)}
\end{cases}
예시는 다음과 같습니다: $`\mathcal{R}_{EE}`$:
Biopsy $`\rightarrow`$confirms$`\rightarrow`$ TumorType,
$`\mathcal{R}_{EA}`$:
CT Scan $`\rightarrow`$visualizes$`\rightarrow`$ Pancreatic Mass,
$`\mathcal{R}_{AA}`$:
HER2 Status $`\rightarrow`$determines$`\rightarrow`$ Trastuzumab Eligibility.
후보 생성: 서술문 $`x`$에 대해 후보 관계 $`\mathcal{T}_{\text{rel}} = \{(h_i, p_i, t_i)\}`$는 Gemini를 통해 생성됩니다:
\mathcal{T}^{\text{gen}}_{\text{rel}} = f_{\text{Gemini}}(\pi_{\text{rel}}(x))
여기서 $`h_i, t_i \in \mathcal{E} \cup \mathcal{A}`$이고 $`p_i \in \mathcal{V}_{\text{verb}}`$입니다.
의미 검증: 각 트리플 $`\tau_i`$는 GPT-4.o에 의해 맥락적 추론을 사용하여 점수화됩니다:
J(\tau_i) = f_{\text{GPT-4.o}}(\pi_{\text{judge}}(\tau_i, x)) \in [0,1]
적대적 필터링: Grok 3은 각 관계를 변형하고 모순을 식별합니다:
\xi(\tau_i) = \frac{|\{\tau_i' \in \mathcal{A}(\tau_i)\ |\ \texttt{contradictory}\}|}{|\mathcal{A}(\tau_i)|}
최종 집합: 수용된 관계 집합은 …