구조-상태 결합 학습으로 네트워크 진화 히스토리 추론
📝 원문 정보
- Title: Inferring Network Evolutionary History via Structure-State Coupled Learning- ArXiv ID: 2601.02121
- 발행일: 2026-01-05
- 저자: En Xu, Shihe Zhou, Huandong Wang, Jingtao Ding, Yong Li
📝 초록
네트워크의 진화 역사로부터 한 개의 최종 스냅샷과 제한된 시간 주석을 추론하는 것은 기본적이면서도 도전적인 과제이다. 현재 접근 방식은 대부분 위상만에 의존하는데, 이는 종종 부족하고 잡음을 포함한 단서를 제공한다. 본 논문은 특정 동적 과정 하에서 수렴된 노드 상태인 네트워크 정상 상태 동역학을 추가적인 널리 접근 가능한 관찰로 활용하여 네트워크 진화 역사 추론에 사용한다. 우리는 CS$^2$를 제안하며, 이는 구조-상태 결합을 명시적으로 모델링하여 위상이 정상 상태를 어떻게 조절하는지와 두 신호가 엣지 판별을 어떻게 공동으로 향상시키는지를 포착한다. 여섯 개의 실제 시간 네트워크에서 여러 동적 과정 하에 평가한 실험 결과 CS$^2$는 강력한 베이스라인을 일관되게 능가하며, 평균 4.0%의 엣지 순위 판별 정확도 향상과 평균 7.7%의 전역 순서 일관성(Spearman-$ρ$) 향상을 보여준다. CS$^2$는 또한 클러스터 형성, 차수 이질성, 허브 성장과 같은 거시적 진화 경로를 더욱 신뢰성 있게 복원한다. 게다가, 정상 상태만을 사용하는 변형은 신뢰할 수 있는 위상이 제한적인 상황에서도 경쟁력을 유지하며, 정상 상태가 진화 추론을 위한 독립된 신호임을 강조한다.💡 논문 해설
1. **기본적인 이해**: 이 논문은 사회적 네트워크, 도시 시스템, 생태학적 네트워크 같은 복잡한 시스템에서 네트워크의 진화 과정을 재구성하는 방법에 대한 연구입니다. 기본적으로, 우리는 시간이 흐름에 따라 변하는 연결을 단순히 한 순간의 스냅샷으로만 보는 것이 아니라, 이 연결들이 어떻게 형성되었는지 이해하려고 합니다. 2. **중급 이해**: 논문에서는 네트워크의 구조와 그 위에서 일어나는 동적 과정(예: 정보 확산)을 결합한 학습 방법론인 Coupled Structure–State (CS$`^2`$)를 제안합니다. 이를 통해, 우리가 네트워크가 어떻게 진화했는지를 더 잘 이해할 수 있습니다. 3. **고급 이해**: CS$`^2`$는 네트워크의 마지막 스냅샷과 그에 따른 정상 상태(예: 정보 확산이 안정된 상태)를 결합하여, 각 연결이 언제 형성되었는지 예측하는 방법을 제안합니다. 이 방식은 기존의 시간 정보가 부족하거나 불완전한 경우에도 네트워크 진화의 역사를 추론할 수 있도록 도와줍니다.📄 논문 발췌 (ArXiv Source)
복잡한 네트워크, 네트워크 진화, 네트워크 재구성, 안정 상태 동력학.
서론
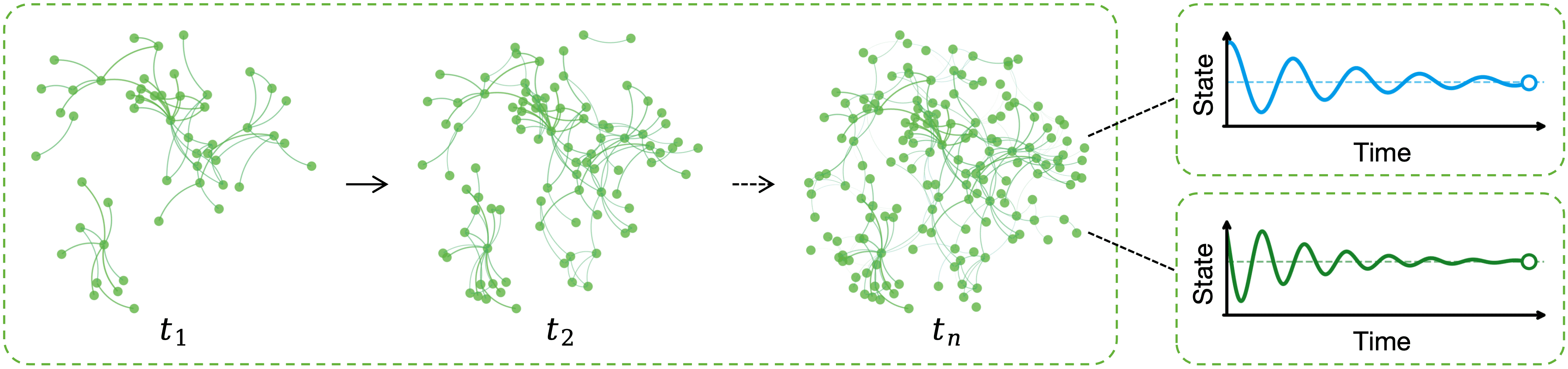
복잡한 시스템, 예를 들어 사회적 네트워크, 도시 시스템, 생태학적 네트워크에서는 네트워크 구조가 지속적으로 진화하지만 관찰은 종종 특정 시간에 단일 스냅샷으로 제한된다(Figure 1). 이 스냅샷은 관찰되지 않은 형성 사건의 연속적인 결과이며, 네트워크의 진화 역사, 핵심 상호작용 및 생성 메커니즘을 암시적으로 인코딩한다. 최종 스냅샷에서 이러한 역사를 추론함으로써 기전적 이해를 진전시키고 영향력의 원천 식별, 기능적 조직 설명, 인과 의존성 분리와 같은 하류 분석을 지원할 수 있다. 그러나 최종 토폴로지는 전체 역사가 단일 정적인 형태로 압축되어 있어 이러한 스냅샷에서 역사를 추론하는 것은 본질적으로 어려운 작업이다.
최근 연구는 이 문제를 최종 토폴로지만을 사용하여 해결하기 시작했다. StructEvo 는 타임스탬프가 있는 몇 개의 연결로부터 구조-시간 매핑을 학습하고 동일 네트워크 내의 나머지 연결을 순위화한다. TopoDiff 는 이 패러다임을 보지 못한 네트워크로 확장하여 여러 시변 네트워크를 통해 훈련함으로써 전이성을 개선하지만, 원래의 내부 네트워크 복원 작업에서 명확한 정확도 향상을 보여주지 않는다. 더 광범위하게 보면, 토폴로지만을 사용하는 접근법은 추론 시 완전하고 신뢰할 수 있는 구조를 가정한다; 실제로 네트워크 관찰이 잡음이 많거나 불완전하거나 심지어 이용 불가능할 경우, 이는 타당성을 제한하고 토폴로지를 넘어서 보충적인 신호를 동반하게 만든다.
토폴로지를 넘어 우리는 안정 상태 동력학을 진화 재구성의 보완적 관찰로 고려한다. 안정 상태는 주어진 동역학 과정 아래에서 수렴된 노드 상태 구성이며, 최종 의견 프로필, 균형 확산 농도 또는 정상 교통 흐름과 같은 예를 들 수 있다. 기본 구조에 대한 동역학적 반응으로서 안정 상태는 토폴로지와 동력학 모두에 의해 공동으로 형성되므로, 최종 스냅샷에서 명시되지 않은 비 국소 종속성을 포착할 수 있다. 안정 상태는 세 가지 이유로 정보를 제공한다. (i) 그들은 네트워크 상호작용의 전반적인 투영을 제공하며, 국부적 지표를 넘어서 고차 구조 패턴에 민감하다. (ii) 서로 다른 연결 형성 순서는 수렴으로 이끄는 서로 다른 궤적을 유발하고 관찰된 안정 상태 내에서 형성 시간과 관련된 쌓인 흔적을 남긴다. (iii) 많은 시스템에서는 연결 형성이 상태에 의해 주도되는 경우가 많다(예: 사회적 네트워크의 동질성 및 생물학적 네트워크에서 표현에 따른 규제), 이는 안정 상태와 진화 순서 간의 내재적인 결합을 생성한다. 또한, 안정 상태는 실용적이다; 그들은 종종 로그, 모니터링 또는 집계 통계로부터 이용 가능하며, 기본 상호작용 네트워크가 잡음이 많거나 불완전하거나 관찰할 수 없더라도 그렇다. 이는 안정 상태를 활용하여 시간 복원을 개선하고 구조 제한 조건 하에서 추론을 가능하게 하는 동기를 제공한다.
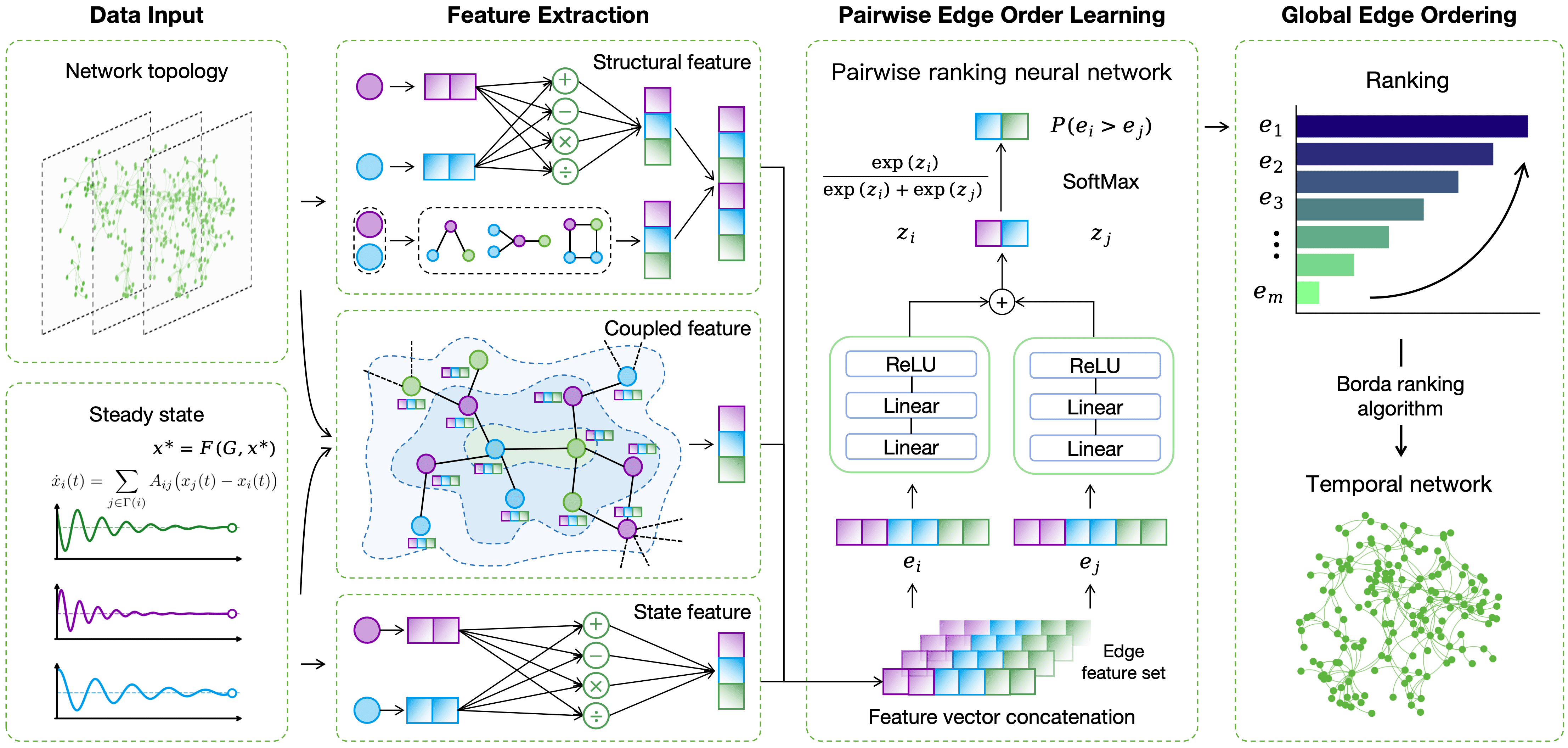
본 논문에서는 일부 연결 시간 스탬프만이 이용 가능한 경우 전체 연결 형성 순서를 복원하는 방법을 연구한다. 우리의 핵심 아이디어는 마지막 스냅샷에 관련된 안정 상태를 보충하고, 일시적 순서 지정을 위한 구조-상태 결합 표현을 학습하는 것이다. 이렇게 하면 모델이 토폴로지적 단서와 동일한 구조가 유발한 동역학적 반응 모두를 활용할 수 있다. 우리는 CS$`^2`$, 동적 정보를 고려한 학습 프레임워크를 제안하며, 이는 쌍별 연결 비교를 통해 일시적 순서 지정을 수행하고 Borda 집합을 통해 전역 시퀀스를 재구성한다. CS$`^2`$는 각 후보 연결에 대한 구조적 및 안정 상태 특성을 추출하고 두 모달리티를 명시적으로 결합하여 순서 지정을 위한 연결 차별화를 개선한다. 실험은 구조와 안정 상태를 결합하면 네트워크 진화 역사 추론에 일관된 성능 향상을 제공함을 보여준다. 특히, 신뢰할 수 있는 구조가 불완전하거나 획득하기 어려운 경우에도 안정 상태 신호는 여전히 경쟁력을 갖추고 있다는 것은 안정 상태가 시간 차별적 단서를 독립적으로 제공하며 구조 제한 조건 하에서 실용적인 추론을 가능하게 한다는 것을 시사한다.
우리의 주요 기여는 다음과 같이 요약된다:
- 우리는 네트워크 진화 역사 추론을 위한 보완적이고 널리 이용 가능한 신호로서 안정 상태 동력학을 도입하고, 왜 안정 상태가 시간 차별적 정보를 포함할 수 있는지 원칙적으로 설명한다. 또한 우리가 보여주는 바와 같이 신뢰할 수 있는 구조가 불완전하거나 이용 불가능한 경우에도 안정 상태 신호는 여전히 경쟁력이 있다는 것을 보인다.
- 우리는 CS$`^2`$, 토폴로지와 안정 상태 간의 상호작용을 명시적으로 모델링하여 순서 지정을 위한 연결 차별화를 개선하는 구조-상태 결합 학습 프레임워크를 제안한다.
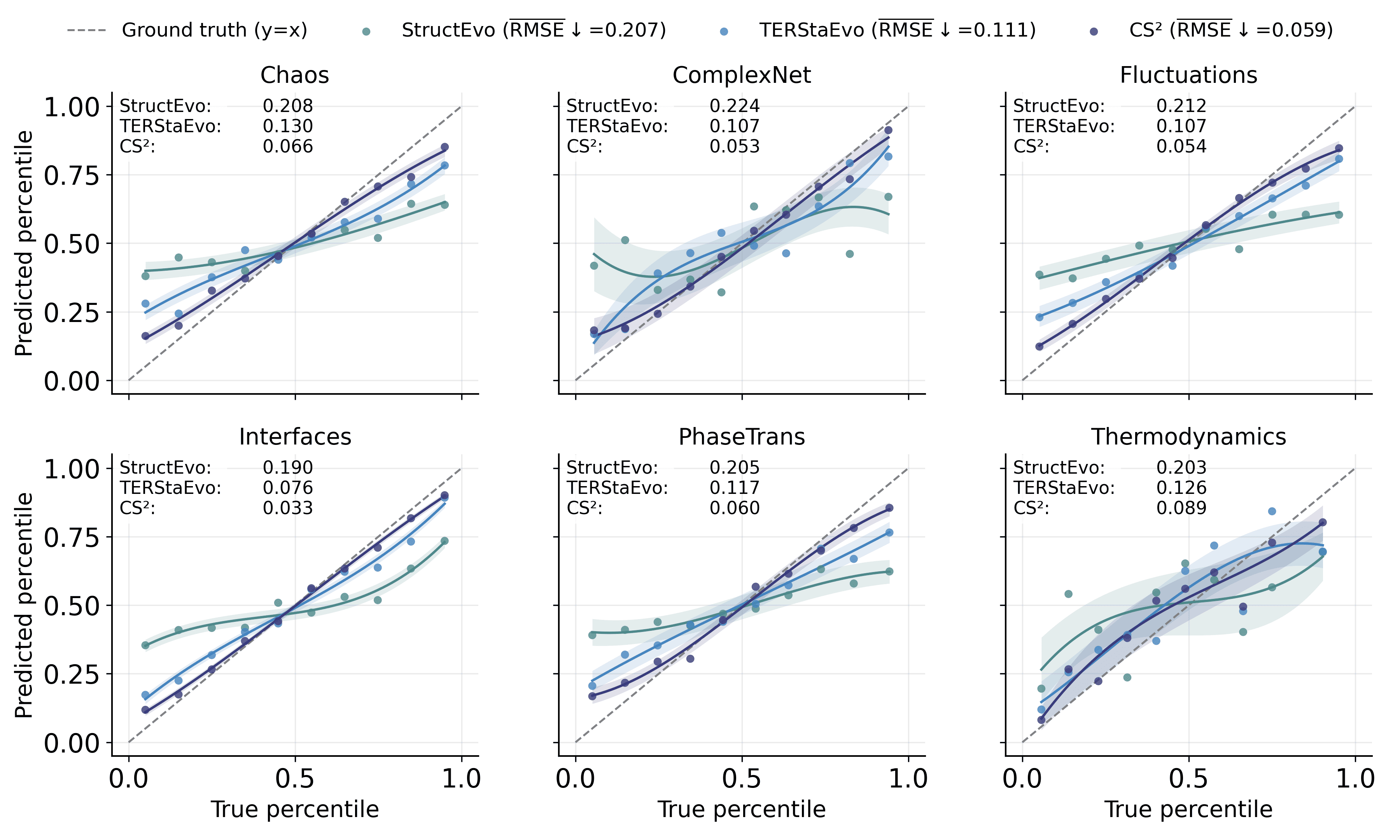
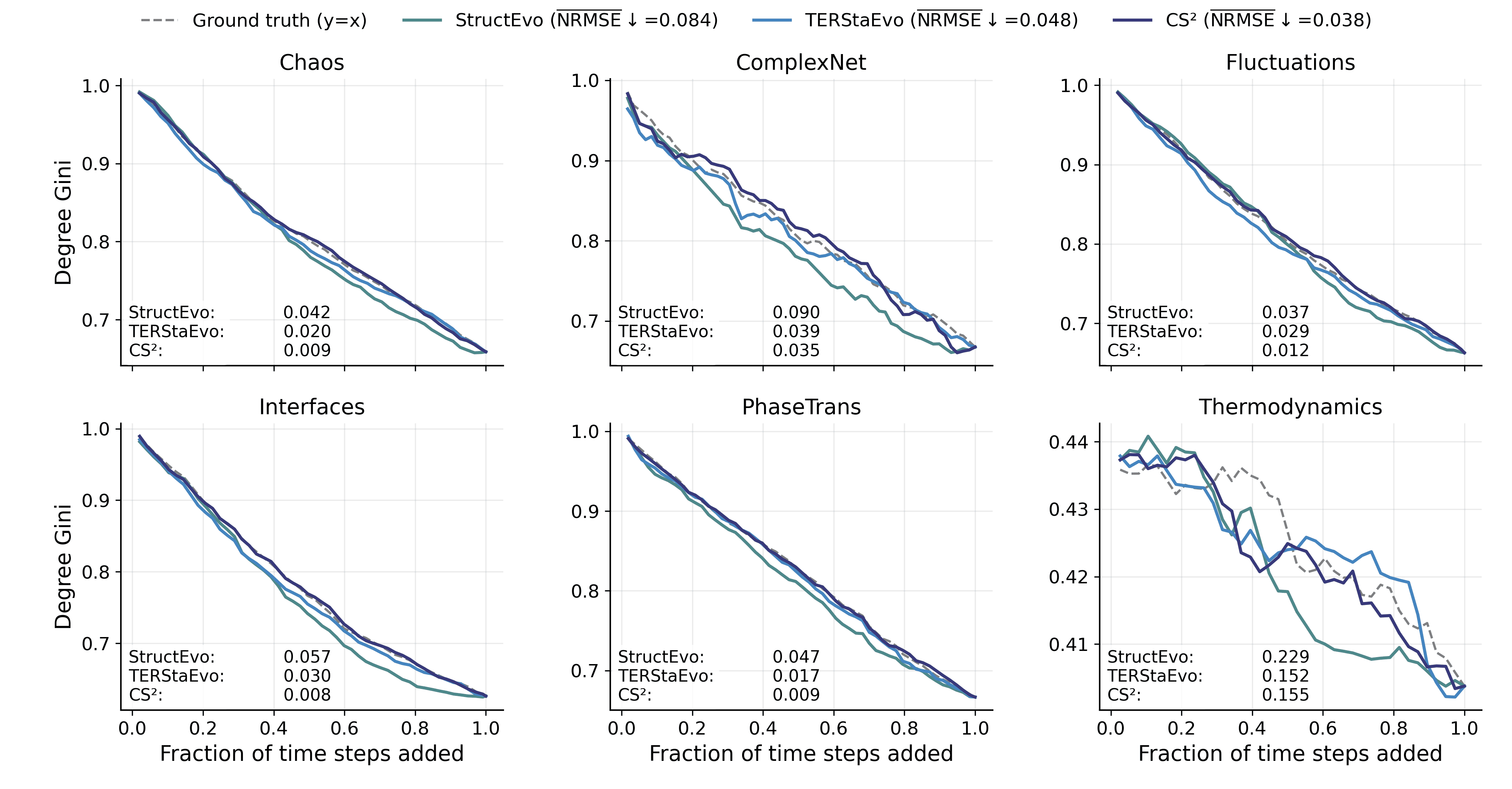
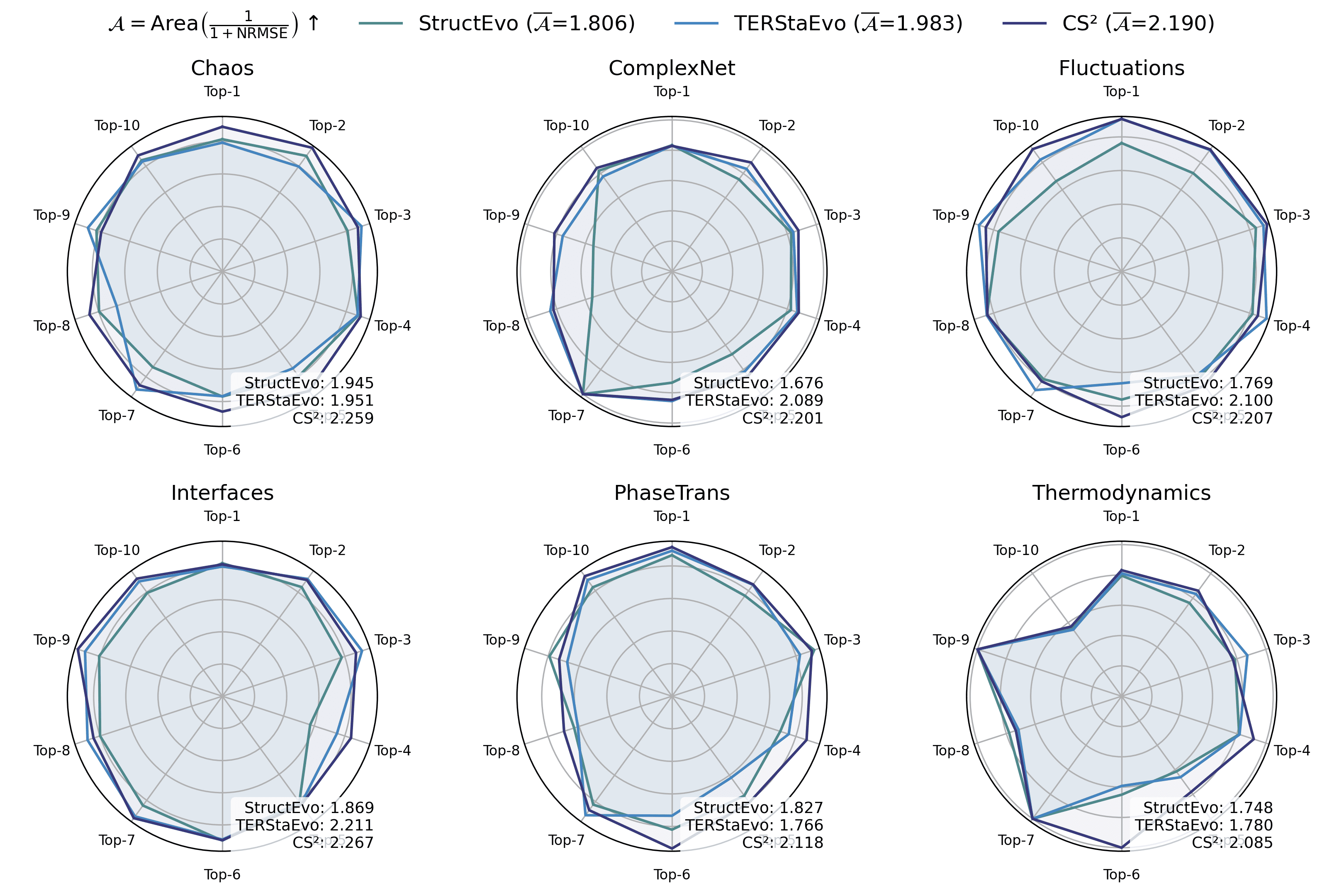
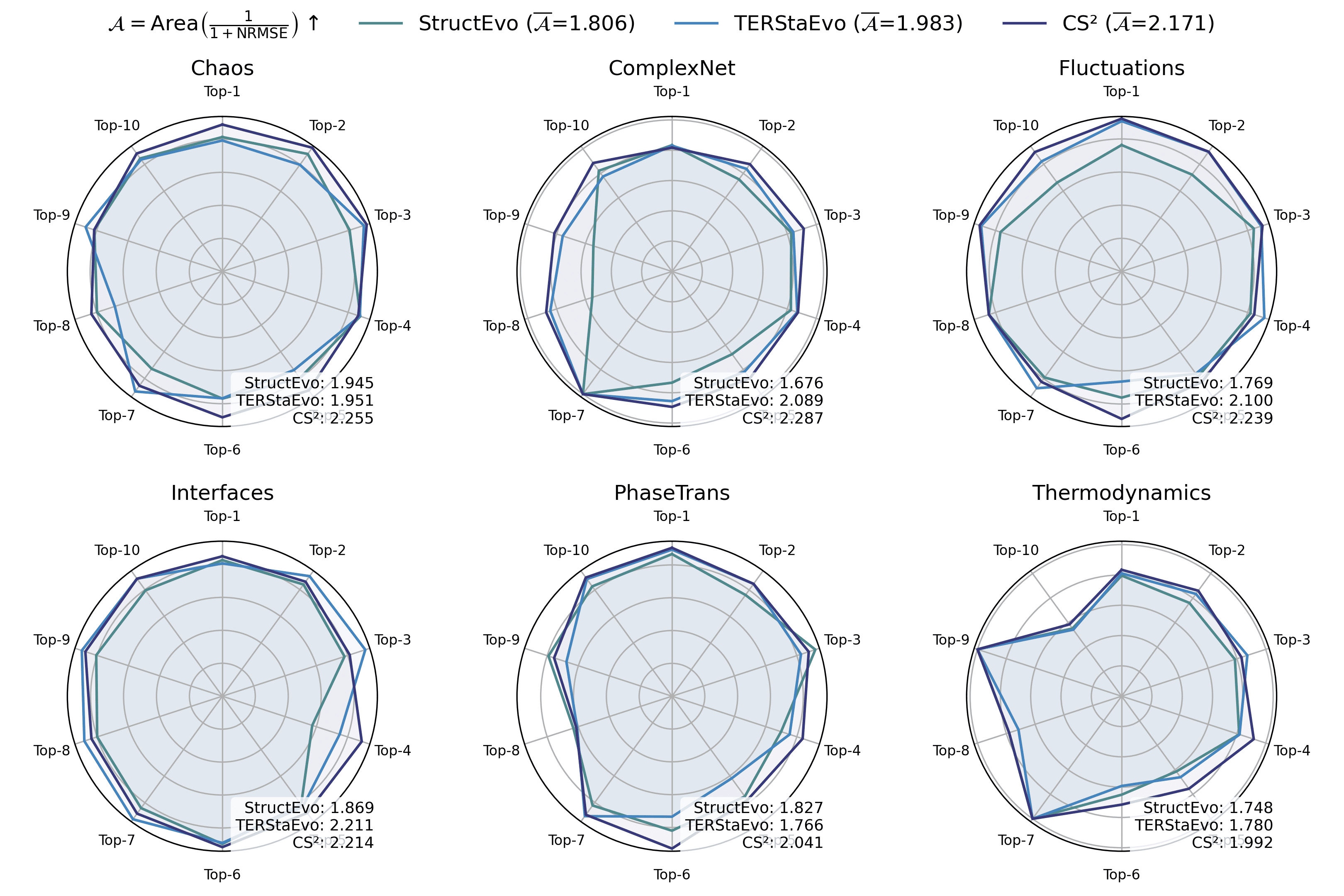
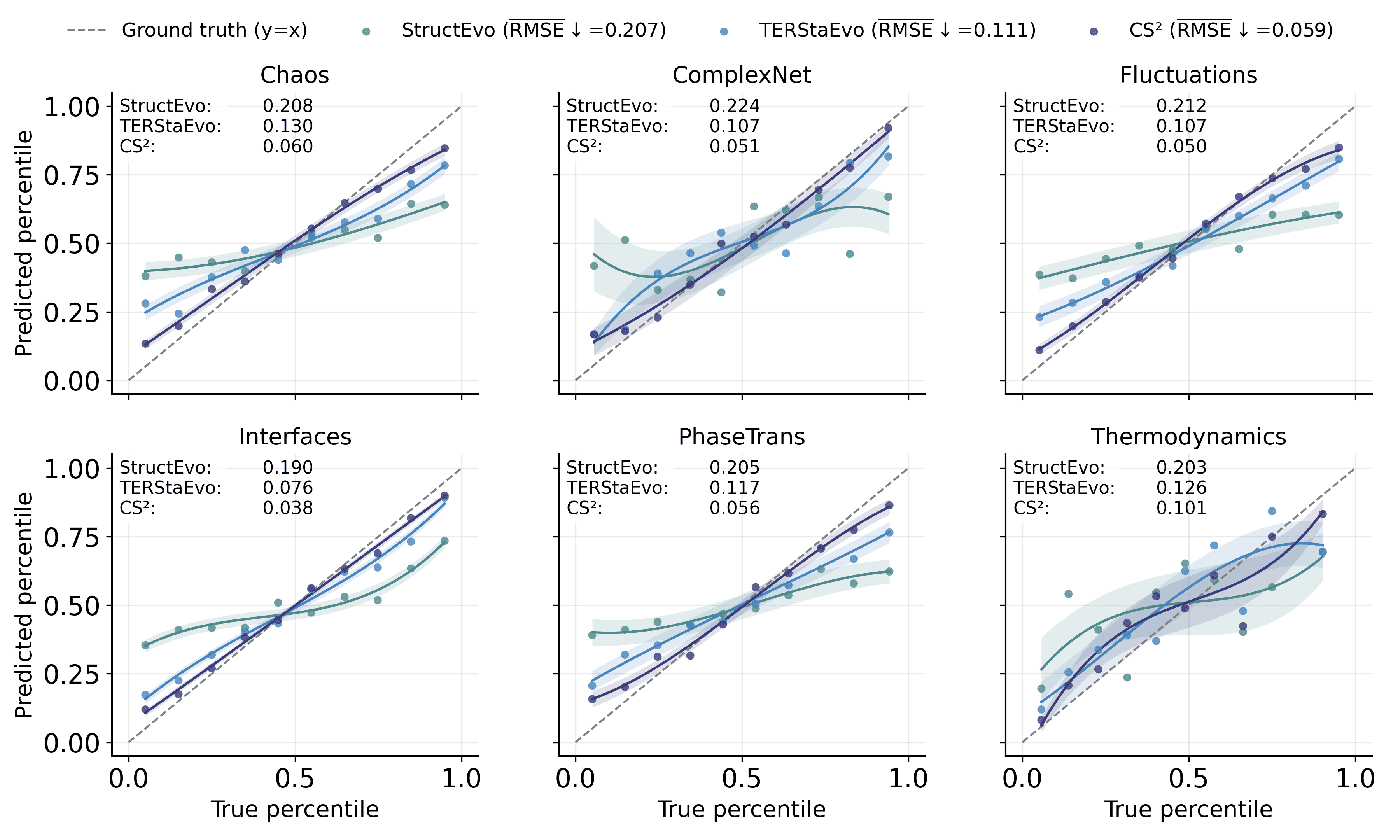
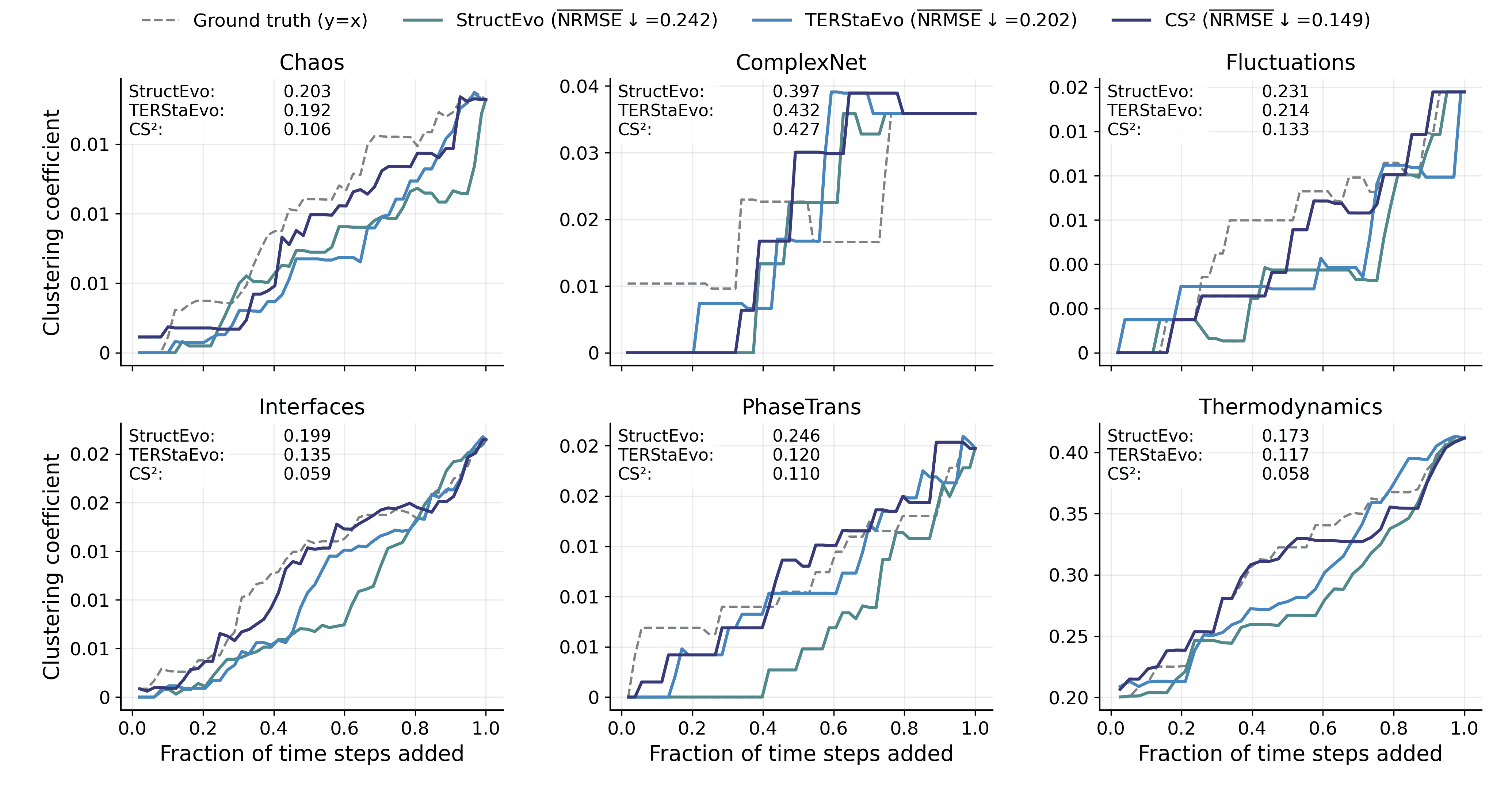
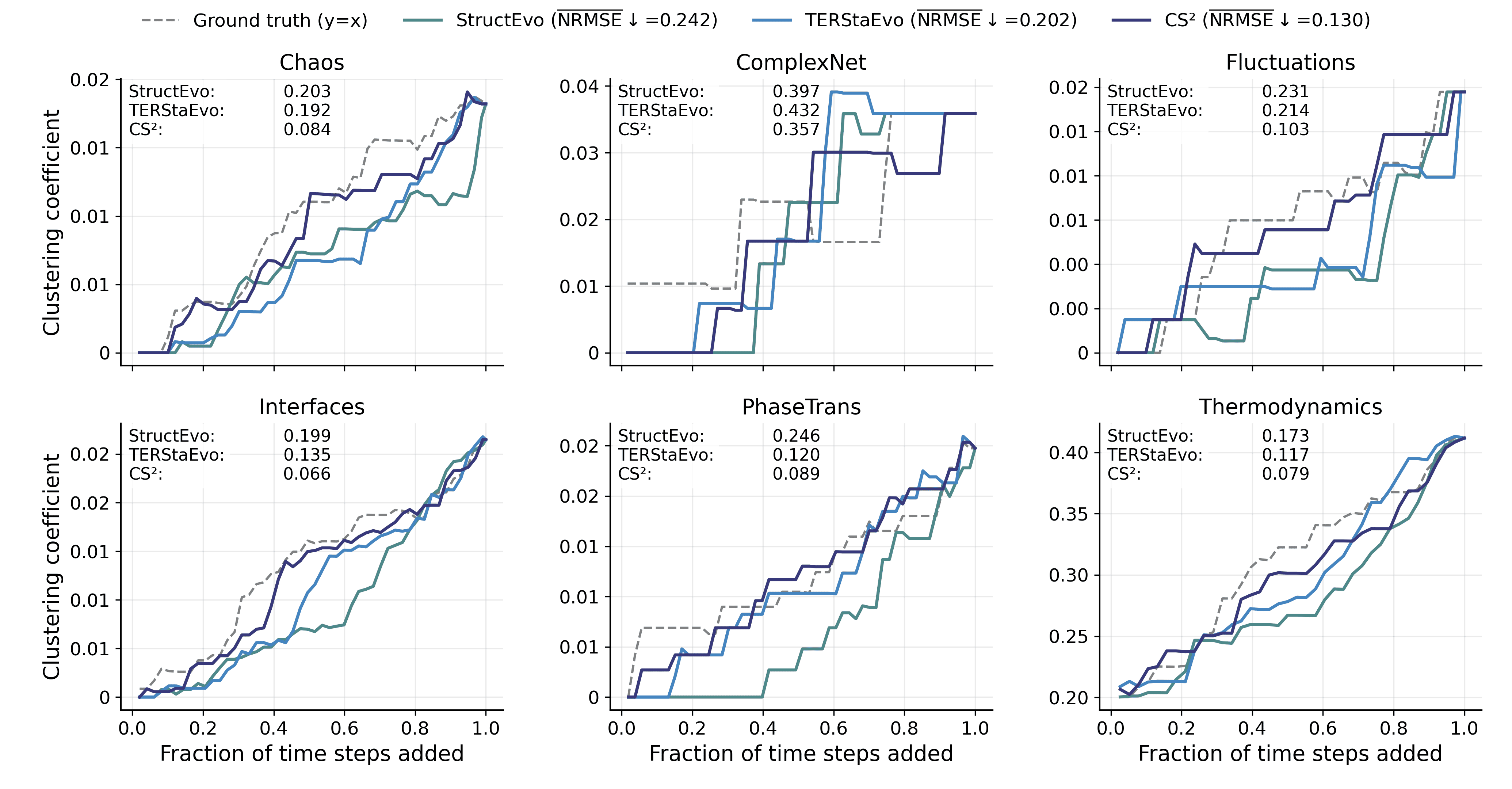
- CS$`^2`$는 쌍별 및 전역 순서 복원에서 일관된 향상을 보여준다. 쌍별 연결 우선순위 정확도는 최대 경쟁 기반 라인에 비해 4.0% 향상되었으며, 전역 순서 일관성(Spearman-$`\rho`$)은 평균적으로 7.7% 개선되었다. 또한 클러스터링 형성, 차수 이질성 및 핵 성장과 같은 거시적 진화 궤도 복원도 더욱 향상되었다.
관련 연구
시변 네트워크
시변 네트워크에 대한 연구는 여러 설정에서 이루어졌지만 대부분의 작업은 여기서 공부하는 역 문제에는 맞춰져 있지 않다—단일 최종 스냅샷과 희소한 시간 정보로부터 존재하는 연결의 형성 순서를 복원하는 것.
클래식 라인은 네트워크 진화를 생성 메커니즘을 통해 모델링하며, 노드와 연결에 대한 성장 규칙을 제시한다. 대표적인 예로는 선호적 첨부(Barabási–Albert) , 소규모 세계 재연결(Watts–Strogatz) , PSO 모델 및 적합도 기반 모델 이 있다. 이러한 모델은 거대한 통계량(예: 차수 분포, 클러스터링, 커뮤니티 구조)이 어떻게 발생하는지 설명하지만, 관찰된 네트워크에서 실제로 형성된 연결 순서를 복원하기보다 시뮬레이션 및 합성을 목적으로 설계되었다.
또 다른 주요 라인은 완전히 관찰 가능한 시변 상호작용 스트림에 대한 딥러닝 방법을 개발한다. 예로는 TGAT , TGN , DySAT 및 EvolveGCN 가 있다. 이러한 방법들은 타임스탬프된 스트림이나 스냅샷의 시퀀스를 입력으로 가정하고 시간 주의력, 시간 인코딩, 순환 업데이트 또는 메시지 패싱을 통해 진화하는 표현을 학습하며, 주로 미래 구조(예: 앞으로 발생할 연결)나 상태 예측에 초점을 맞춘다. 반면, 우리의 설정은 오직 단일 최종 스냅샷과 누락된 시간 스탬프를 제공하며, 목표는 존재하는 연결의 역사적 순서를 추론하기보다 미래 이벤트를 예측하는 것이 아니다. 이러한 관찰성 및 목적에 대한 근본적인 차이로 인해 시변 스트림 모델은 정적 스냅샷에서 진화 재구성을 직접 적용할 수 없다.
우리의 설정과 가장 가까운 것은 부분적인 시간 스탬프 지도 하에서 연결 순서를 추론하는 학습 기반 진화 재구성 방법이다. StructEvo (Nature Communications)는 소수의 레이블된 연결로부터 구조-시간 매핑을 학습하고 최종 스냅샷 내의 국부적 패턴을 사용하여 동일 네트워크 내의 나머지 연결 순위화한다. TopoDiff 는 이 패러다임을 보지 못한 네트워크로 확장하며 시뮬레이션 시간 증강을 통해 여러 시변 네트워크에서 훈련함으로써 교차 네트워크 전이성을 개선한다.
이런 진전에도 불구하고, 토폴로지만을 사용한 재구성은 실제로 제한적이다. 최종 스냅샷은 연결성을 인코딩하지만, 종종 엣지 형성과 함께 동반되는 동력학적 반응이나 상태에 따른 메커니즘을 인코딩하지 않으며 이는 복잡한 시스템에서 식별 가능성을 감소시킨다. 또한 정확한 토폴로지는 획득하기 비싸고 잡음이 많거나 불완전할 수 있어 순수하게 토폴로지에 기반한 추론을 덜 신뢰성 있게 만들며, 구조를 넘어서 보충적 관찰 신호를 동반하게 만든다.
네트워크 위의 안정 상태 동력학
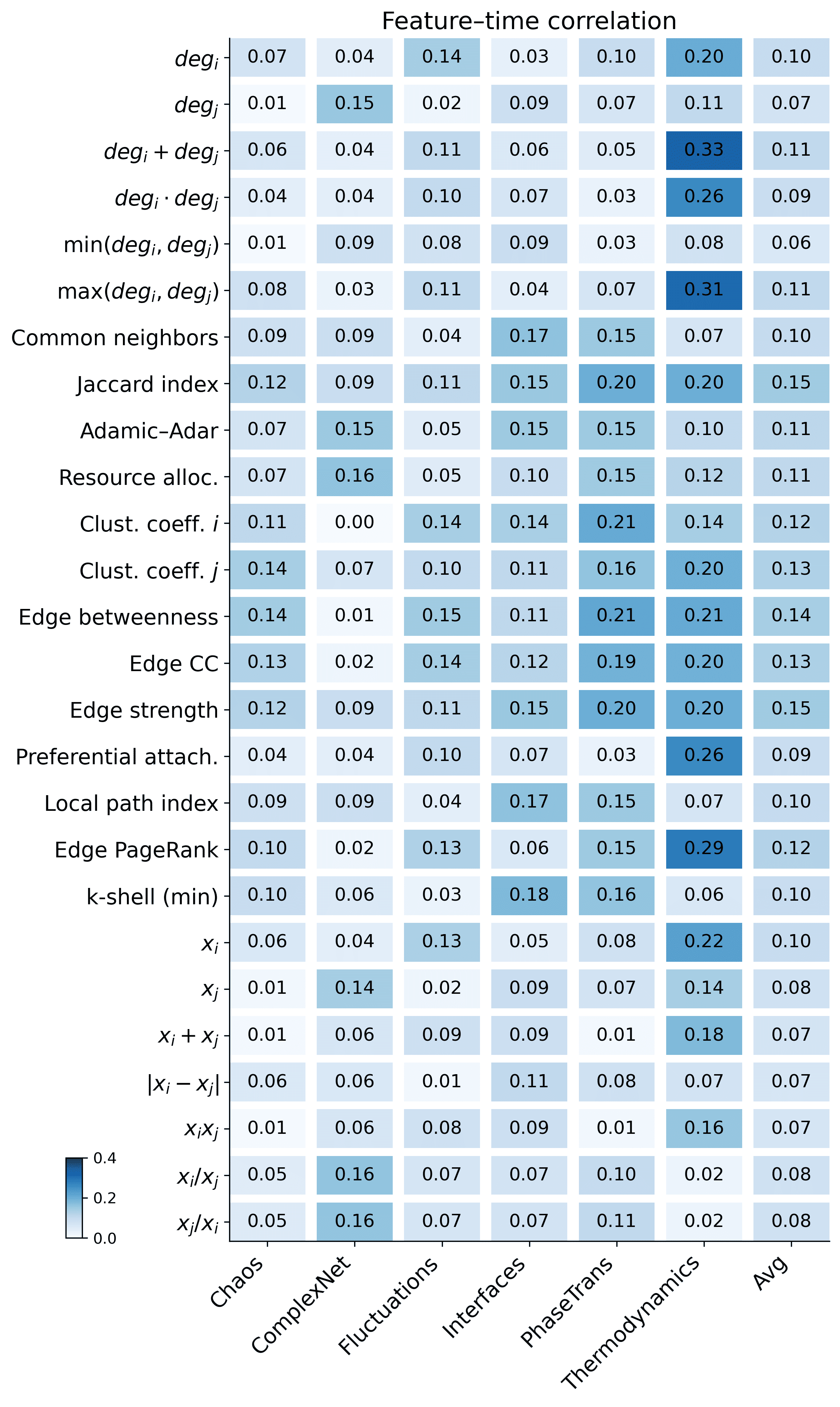
네트워크 동역학은 정보, 의견 또는 상태가 그래프에서 어떻게 진화하는지를 이해하기 위한 잘 확립된 렌즈를 제공한다. 대표적인 과정에는 확산 , 무작위 걷기 , 동조화 , 의견 동역학 및 전염병 확산 이 있다. 고정된 토폴로지 아래에서 이러한 동역학은 종종 안정 상태에 수렴하며 이는 그래프 구조와 동력학 규칙 모두에 의해 공동으로 결정된다. 여러 단계의 상호작용을 네트워크 전체에서 집약화하므로, 안정 상태는 국부적 구조 지표를 보완하는 전반적인 토폴로지 반응으로 해석될 수 있다.
이전 연구는 안정 상태와 관련된 확산 균형량이 고차 구조 속성, 커뮤니티 조직 , 중앙성 이질성 및 전역 연결 패턴 을 포함하여 반영한다는 것을 보여주었다. 이러한 관점은 또한 표현 학습과 스펙트럼 그래프 이론에서 널리 사용된다: 열커널 임베딩 , 확산 상태 거리 , 무작위 걷기/확산 기반 임베딩 및 라플라시안 스펙트럼 분해 는 수렴 행태를 활용하여 일회성 토폴로지 설명자에서 어렵게 복원할 수 있는 전역 유사성과 은닉 구조 패턴을 포착한다.
그들의 구조 특성화 및 임베딩 학습에 널리 사용되었음에도 불구하고, 안정 상태는 정적 스냅샷에서의 시변 추론을 위한 관찰 신호로서 거의 탐구되지 않았다. 특히 어떻게 안정 상태 관찰을 활용하여 연결 형성 순서를 복원하고 네트워크 진화 역사 재구성을 할 수 있는지 여전히 크게 개방된 문제이다. 이 간극은 본 논문에서 제안하는 안정 상태에 기반한 진화 재구성 프레임워크를 동기 부여한다.
예비 지식
쌍별 비교를 통한 네트워크 진화 재구성
각 연결에 대한 절대 형성 시간을 직접 예측하는 것은 종종 필요하지 않고 진화 재구성을 불안정하게 만든다. 연결 시간은 연속적이며 일반적으로 같은 네트워크 내에서만 비교 가능하며, 상대적 순서가 주요 관심 대상이다. 부분적인 시변 지도 하에서는 시간 회귀가 잡음에 민감하고 레이블의 희소성에 민감할 수 있다.
대신 우리는 쌍별 시간 우선순위를 예측한다: 두 개의 연결을 주어졌을 때, 어느 것이 먼저 형성되었는지를 결정한다. 이는 원래 회귀 문제를 최적화하기 더 쉽고 희소한 레이블 하에서 더욱 견고한 이진 분류 문제로 변환한다. 쌍별 관계가 가능해지면, 이를 모든 연결에 대한 완전한 순서로 집합할 수 있다.
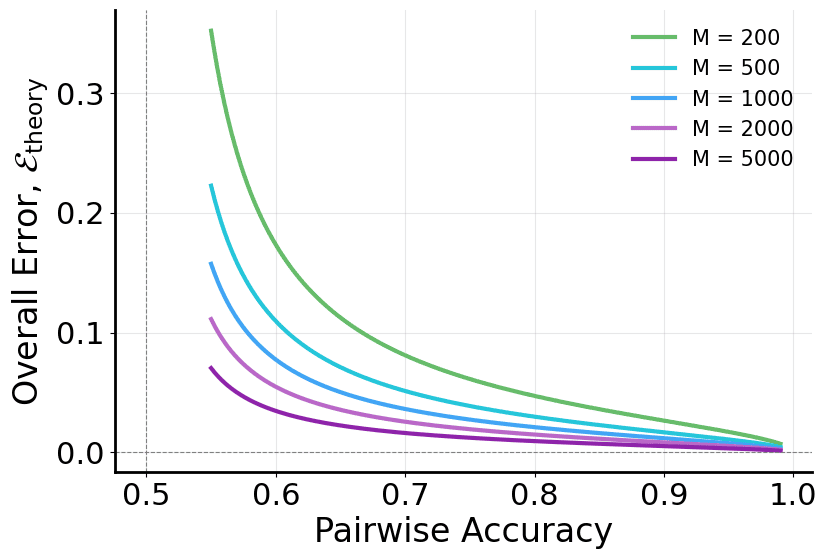
우리는 또한 로컬 쌍별 정확도와 전역 순서 품질 간의 양적 연관성을 설정한다. $`p`$를 무작위로 샘플링된 쌍에 대한 우선순위 예측을 정확히 하는 확률이라고 하자. 그러면 회수된 전역 순서의 기대 오차는
\begin{equation}
\mathcal{E}_\text{theory} =
\sqrt{\frac{p(1-p)}{(2p-1)^2}}
\frac{1}{\sqrt{M}},
\label{eq:equivalence}
\end{equation}에서, $`M`$은 연결의 수를 나타낸다. 완전한 유도는 부록 6.1에 제공된다.
쌍별 우선순위 예측을 얻은 후, 우리는 Borda 계산을 통해 전역 순서를 재구성한다. 각 연결에는 “승리” 수와 동일한 점수가 할당되며, 예측된 비교와 가장 일치하는 순서가 반환된다. 세부 사항은 알고리즘 [alg:borda_reconstruction]에 제공된다.
요약하자면, 쌍별 학습은 연결 형성 순서를 복원하는 목표와 더 잘 일치하며 네트워크 진화 재구성을 위한 최적화 친화적인 경로를 제공한다. Figure 2은 Eq. [eq:equivalence]에 의해 암시되는 쌍별 정확도와 회수된 전역 순서의 기대 오차 간의 이론적 연결을 시각화한다.
문제 정의
목표 시간에 완전히 관찰된 무향 네트워크를 고려한다.
G=(V,E),\qquad |V|=N,\ |E|=M,여기서 $`E`$의 엣지들은 알려지지 않은 과정에 의해 순차적으로 생성된다. 각 연결 $`e\in E`$, 우리는 그 정확한 형성 순서를 다음과 같은 정규화된 시간으로 나타낸다.
\alpha_e \in [0,1],여기서 작은 값은 더 이른 형성을 나타낸다.
전체 연결 중 일부만이 관찰 가능한 형성 시간을 가진다. 이를 레이블 세트로 표시한다.
E_{\mathrm{lab}}=\{e_i \mid \alpha_{e_i} \ \text{known}\},그리고 나머지 연결은 레이블되지 않은 상태이다.
E_{\mathrm{unk}} = E \setminus E_{\mathrm{lab}}.토폴로지를 넘어서, 우리는 동역학 과정(예: 확산, 유전자 조절 또는 의견 동역학)을 $`G`$에 적용하여 수렴할 때까지 실행한 안정 상태 벡터를 관찰한다.
\mathbf{x} = (x_1, x_2, \ldots, x_N),여기서 $`x_i`$는 노드 $`i`$의 안정 상태 값이다. 안정 상태는 구조 $`G`$와 동역학 규칙 모두에 의해 공동으로 결정되며, 이는 동력학 아래에서 토폴로지의 전반적인 반응으로 볼 수 있다.
$(G,\mathbf{x})$와 부분적 시간 레이블 $`E_{\mathrm{lab}}`$, 우리의 목표는 완전한 연결 형성 순서를 재구성하는 것이다. 구체적으로 다음과 같이 정의한다:
1. 쌍별 기본 우선순위. 임의의 두 개의 연결 $`e_i, e_j \in E`$에 대해 그들의 기본 우선순위 관계를
R(e_i,e_j)=
\begin{cases}
1, & \alpha_{e_i} < \alpha_{e_j},\\[2mm]
0, & \alpha_{e_i} \ge \alpha_{e_j}.
\end{cases}여기서 $`R(e_i,e_j)=1`$은 $`e_i`$가 $`e_j`$보다 먼저 형성된 것을 나타낸다.
2. 쌍별 순서 예측. 우리는 결정 함수
\widehat{R}(e_i,e_j)=f\big(G,\mathbf{x},e_i,e_j\big),을 학습하여 임의의 연결 쌍에 대한 우선순위를 예측한다.
3. 전역 순서 재구성. 쌍별 예측 $`\widehat{R}(e_i,e_j)`$에서, 우리는 Borda 집합(이 논문에서는 이를 사용)을 통해 로컬 비교와 가장 일치하는 전역 순서를 구축한다.
\hat{\pi}: E \rightarrow \{1,2,\ldots,M\},여기서 $`\hat{\pi}(e)`$는 재구성된 시퀀스에서 연결 $`e`$의 위치를 나타낸다.
예측 진화 시퀀스는 다음과 같이 쓸 수 있다.
\hat{T} = \{e_{k_1}, e_{k_2}, \ldots, e_{
</div>
<div style="margin-top: 20px;"><a href="https://arxiv.org/pdf/2601.02121.pdf" target="_blank">ArXiv 원문 PDF 보기</a></div>
<div class="mobile-ad w-full my-6 text-center" style="border: 2px dashed red; background: #ffe6e6;"><ins class="adsbygoogle" style="display:block" data-ad-client="ca-pub-1873718820012422" data-ad-slot="auto"></ins><script>(adsbygoogle = window.adsbygoogle || []).push({});</script></div>
<br>
<h4>📊 논문 시각자료 (Figures)</h4>
<br>
<br>

<br>
<br>
<br>
<br>

<br>
<br>
<br>
<br>
<br>
<br>
<br>
<br>
<br>
<br>

<br>
<br>
<br>
<br>
<br>
<br>

<br>
<br>
<br>
<br>
<br>
<br>

<br>
<br>
<br>
<br>

<br>
<br>

<br>
<br>

<br>
<br>

<br>
<br>

<br>
<br>
<h4 style="margin-top: 3rem; margin-bottom: 1rem; border-bottom: 1px solid #e5e7eb; padding-bottom: 0.5rem;">감사의 말씀</h4>
이 글의 저작권은 연구하신 과학자분들께 있으며, 인류 문명 발전에 공헌해주신 노고에 감사를 드립니다.